Reemplazar a Matlab con Python - Parte 2: Formateo de subtramas e indexación de datos
Usé Python para completar la primera parte de la tarea de mi publicación anterior en la serie. Aprendí sobre formatear gráficos e indexar los marcos de datos de Pandas.
Luis Medina
Hace un par de semanas, publiqué la segunda entrada de la de reemplazo de Matlab con Python que comencé en agosto. Estoy compartiendo ejemplos de cómo hacer eso, con algunos comentarios sobre los problemas que encuentro mientras aprendo Python.
en la publicación anterior de la serie, realmente no compartí ningún código de Python. Era un ejemplo de cómo usaría MATLAB para obtener información de los datos sin procesar del autobús CAN CAN de un automóvil de carrera.
En esta entrada, estoy usando Python para hacer la primera parte de la tarea: visualice las señales que tenemos creando una cifra con subtramas, filtrar los datos de aceleración sin procesar, calcular la velocidad del vehículo para los dos conductores y compararla. Me tomó más del doble del tiempo hacerlo en Python que con Matlab. Eso se debe principalmente a que he estado usando Matlab durante muchos años, y me estoy acostumbrando a Python. Especialmente al formatear parcelas, hay varias cosas que manejar de manera diferente a Matlab, o incluso de una biblioteca de Python a otra.
Como siempre, tenga en cuenta que esto no pretende ser un curso de Python, sino una serie de ejemplos prácticos.
¡Muy bien, al código!
Importando los datos
Estoy siguiendo el mismo flujo de trabajo que usé para la versión de Matlab de esto , donde también proporcioné más contexto sobre lo que tenemos. Historia corta, queremos comparar el rendimiento de dos controladores, y tenemos los datos del automóvil para cada uno en archivos CSV separados.
Este es el código con el que escribí para comenzar. Ya hemos visto cómo cargar información de los archivos CSV y usar pandas para manejar nuestros datos.
Al usar Python, lo primero que hacemos es importar las bibliotecas que vamos a usar. No necesariamente tenemos que poner las importaciones encima de todo, pero en general es una buena práctica.
#%% Import libraries
import pandas as pd
import numpy as np
import matplotlib.pyplot as plt
#%% Load data from CSV files
df1 = pd.read_csv('driver1.csv')
print(df1.columns)
df2 = pd.read_csv('driver2.csv')
print(df2.columns)
#%% Define constants
gear_ratio = 16 # Transmission gear ratio
r = 0.263 # Wheel radius [m]
>> Index(['Time_s', 'BrakeFront', 'AccX', 'spd_rpmFL', 'spd_rpmFR', 'spd_rpmRL',
'spd_rpmRR'],
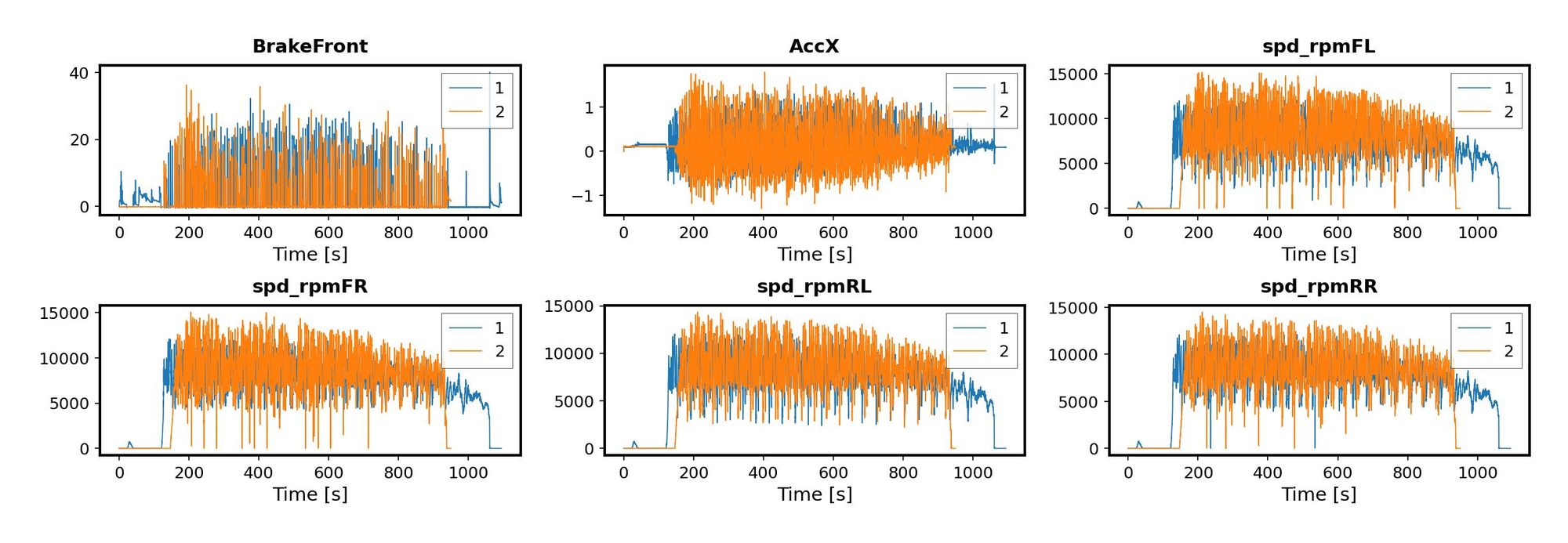
dtype='object') ¡Todo está bien! Tenemos una base de tiempo única y 6 señales en cada archivo. Pero tenemos que asegurarnos de que sean consistentes y se pueden usar juntos. Es por eso que lo siguiente que hice en Matlab fue trazar las variables.
Cuando
intenté hacer lo mismo usando Python, me llevó un tiempo. Más de lo que
me siento cómodo admitiendo. Estaba obstinadamente tratando de obtener
exactamente el mismo aspecto que obtuve con la configuración
predeterminada en Matlab y, obviamente, eso requirió mucho formato. En
algún momento lo obtuve, pero el número de pruebas con opciones de
formato simplemente no valió la pena. Solo mira el plt.rcParams.update
¡Llame al final del siguiente fragmento de código! Mi consejo: si está
reemplazando a Matlab con Python, no intente obtener exactamente el
mismo aspecto en sus gráficos a menos que haya una necesidad real de
eso, en mi caso, no lo había.
# If needed, reset plot parameters from any previous plot
# plt.rcParams.update(plt.rcParamsDefault)
fig = plt.figure(figsize = (1200/300, 600/300), dpi = 300)
c = 1 # subplot counter
for col in df1.columns[1:]:
ax = plt.subplot(3, 3, c)
ax.plot(df1['Time_s'], df1[col], label = '1', linewidth = 0.2)
ax.plot(df2['Time_s'], df2[col], label = '2', linewidth = 0.2)
leg = ax.legend(edgecolor = 'black',
borderpad=.5, borderaxespad = .5,
fontsize = 3, fancybox = False,
loc = 'upper right')
plt.xlabel('Time [s]', fontsize = 3.5)
plt.title( col, fontweight="bold", y = 1.05, fontsize = 3.5)
leg.get_frame().set_linewidth(.1)
c = c + 1
plt.rcParams.update({'axes.grid': False,
'xtick.labelsize': 3,
'ytick.labelsize': 3,
'axes.labelpad': 1.0,
'axes.labelsize': 3.5,
'axes.linewidth': 0.5,
'ytick.major.pad': 1,
'ytick.minor.left': False,
'xtick.minor.bottom': False,
'ytick.major.size': 1,
'ytick.major.width': .2,
'xtick.major.pad': 1,
'xtick.major.size': 1,
'xtick.major.width': .2})
fig.tight_layout()
fig.subplots_adjust(
top=0.92,
bottom=0.1,
left=0.05,
right=0.98,
hspace=0.6,
wspace=0.2
)
fig Algunos de los recursos útiles que encontré en línea para aprender sobre el formato son: en primer lugar, la documentación . En segundo lugar, el innumerable número de sitios con tutoriales de Python en línea, solo busqué en Google mis preguntas y encontró buenas respuestas en la primera página de resultados. Tercero, preguntas previamente respondidas en StackOverflow .
Algo agradable sobre Python es que podemos iterar directamente a través de las columnas de nuestro marco de datos, en lugar de crear un índice y luego cortar el marco de datos por posición de columna. Al hacerlo de esta manera, solo necesitaba hacer un contador para pasar al siguiente gráfico, y los datos se seleccionaron de la columna actual. Los resultados no son idénticos a los que obtuve en Matlab, pero está bien.
de la biblioteca de nivel relativamente bajo pyplot y desea obtener un formato realmente específico. Si afloja un poco el formato y se abre a otras opciones, como Sevorn , por ejemplo, puede salirse con la suya con muy poco código para obtener algunas tramas impresionantes. O, si necesita hacer tramas interactivas, podría usar Plotly , como lo hice al visualizar cómo era el lecho de mi impresora 3D . Mostraré otro ejemplo a continuación, cuando comparemos las velocidades de los dos conductores.
Bien, podemos hacer tramas sin estresar demasiado sobre el formato y vivir con él.
Sigamos adelante.
Filtrado de datos de aceleración
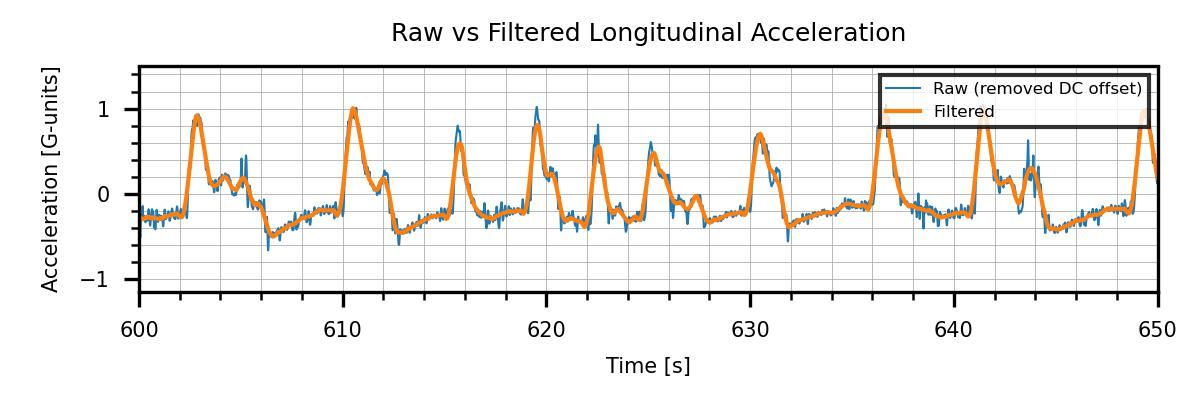
Como en el ejemplo de MATLAB, utilicé un filtro digital para suavizar las señales de aceleración. Estoy filtrando solo aquellos porque son los únicos que contienen datos sin procesar de sensores. Así es como se ven los datos de aceleración en bruto cuando se comparan con el filtrado:
El código que usé para hacer esto es bastante simple.
Primero,
calculé la suma de las velocidades de los cuatro motores en la rueda.
Para hacer esto, necesitaba seleccionar solo las columnas para esas
variables y calcular la suma para cada fila. En MATLAB podemos
seleccionar datos de tablas utilizando índices numéricos y paréntesis
redondo directamente, y el resultado es un objeto de tabla. Con Python,
para indexar un marco de datos de esta manera, necesitamos usar el iloc
método. Esto le permite cortar el marco de datos utilizando índices
numéricos. En este caso, seleccioné todas las filas, y luego las
columnas desde la tercera hasta el final, por lo que el objeto devuelto
también es un marco de datos.
Para calcular la suma de cada fila, utilicé el sum Método de Dataframes, pasando como argumento axis = 1
Para indicar que quiero sumar cada fila y poner los resultados en una
columna. Para los marcos de datos, las filas son el eje 0 y las columnas
son el eje 1. Esto es equivalente al concepto de Matlab de filas y
columnas en tablas. Entonces, cuando sumo a lo largo del eje 1, agrego
todos los valores de la columna en cada fila.
Los resultados se almacenan en nuevas columnas llamadas 'SumSpeeds' en los DataFrames originales.
df1['SumSpeeds'] = df1.iloc[:,3:].sum(axis=1)
df2['SumSpeeds'] = df2.iloc[:,3:].sum(axis=1)# calculando las velocidades del motor ¡Fresco! A continuación, eliminé
las compensaciones de DC de las señales de aceleración. Seleccioné solo
las filas en las que la suma de las velocidades era cero y calculé la
mediana de los valores de aceleración. Luego resté este desplazamiento
distinto de cero de las señales de aceleración. Así es como hice eso:
df1['AccX_NoOffs'] = df1['AccX'] - df1[df1['SumSpeeds'] == 0]['AccX'].median()
df2['AccX_NoOffs'] = df2['AccX'] - df2[df2['SumSpeeds'] == 0]['AccX'].median()# Retire el desplazamiento de DC de las señales de aceleración en bruto En este caso, corté el marco
de datos utilizando un enfoque diferente. Comparé la columna 'SumSpeeds'
con cero, creando un objeto en serie con declaraciones lógicas
(verdaderas/falsas). Esto es equivalente a hacer myseries = df_A['SumSpeeds'] == 0.
Luego, corté los marcos de datos originales usando esa serie como un
índice. Esto devolvió un nuevo marco de datos que contiene todas las
filas en las que se cumple la condición, y todas las columnas, como en df_B = df_A[myseries]. Finalmente, seleccioné la columna 'ACCX' de la marca de datos resultante y calculé la mediana de ella. Habría parecido DC_offset = df_B['AccX'].median(). Y esto es lo que resté de las señales originales.
La última parte fue filtrar las señales corregidas y finalmente hacer la figura que mostré anteriormente, comparando las señales sin procesar y filtradas:
from scipy.signal import savgol_filter
df1.AccX = savgol_filter(df1.AccX_NoOffs, 101, 3)
df2.AccX = savgol_filter(df2.AccX_NoOffs, 101, 3)
# reset figure parameters if previously changed
#plt.rcParams.update(plt.rcParamsDefault)
# Compare raw acceleration vs filtered
fig = plt.figure(figsize = (1200/300, 400/300), dpi=300)
plt.plot(df1.Time_s, df1.AccX_NoOffs, label = 'Raw (removed DC offset)', linewidth = .5)
plt.plot(df1.Time_s, df1.AccX, label = 'Filtered', linewidth = 1)
plt.xlabel('Time [s]')
plt.ylabel("Acceleration [G-units]")
plt.xlim(600, 650)
leg = plt.legend(fancybox = False,
edgecolor = 'inherit', fontsize = 4,
loc = 'upper right')
plt.title('Raw vs Filtered Longitudinal Acceleration')
plt.rcParams.update({'font.size': 5,
'axes.grid': True,
'axes.grid.which': 'both',
'xtick.minor.visible': True,
'ytick.minor.visible': True,
'grid.linewidth': 0.2,
'lines.linewidth': 0.2})
# Display figure
plt.tight_layout()
fig Filtrado de datos de aceleración una vez que se han eliminado las compensaciones de CC Implementé el mismo tipo de
filtro que usé en MATLAB, solo necesitaba una importación de biblioteca
adicional. Sin embargo, los argumentos de entrada para la versión de
Python savgol_filter(data_array, window_size, poly_order) se intercambian con respecto a Matlab (recuerde que usé sgolayfilt(data_array, poly_order, window_size)
en la publicación anterior). Podemos detectar este tipo de cosas
mirando la documentación. ¡Siempre lea la documentación! Con Python,
puede acceder a él rápidamente escribiendo print(<library,object or function>.__doc___) o simplemente help(<library,object or function>) .
Desafortunadamente, la documentación oficial para muchas funciones de Python no es tan detallada como la contraparte de Matlab. Por ejemplo, compare la de Matlab y Python (SCYPI) para el filtro que utilizamos. El primero es mucho más claro. Esto puede ser una gran cosa para muchas personas que desean aprender a Python proveniente de Matlab, ¡pero hay una manera de darlo! Hay muchos más tutoriales en línea sobre Python que para Matlab. Solo tienes que abrazar el poder de buscar en Google y perder el miedo a sitios como StackOverflow . Por ejemplo, encontré este gran artículo sobre el filtro que estamos usando. Además, lo mejor que puede hacer es consultar algún libro de procesamiento de señales para obtener más información sobre los filtros digitales.
¡De acuerdo! Me estoy desviando desde el punto de este artículo. Volviendo a los ejemplos de código.
Encontrar la velocidad del vehículo: indexación de datos de datos
Como mencioné en el ejemplo de Matlab, podemos estimar la velocidad del vehículo utilizando la información de las velocidades de los cuatro motores en la rueda. Solo necesitamos tratar de manera diferente los casos en los que el vehículo estaba acelerando y aquellos en los que estaba frenando. Para hacer esto, necesitaremos indexar (cortar) el marco de datos basado en condiciones similares a las que hice con los datos de aceleración sin procesar al calcular el desplazamiento de DC. Vea cómo lo hice en el siguiente código:
import timeit
%%timeit
df1['Vehicle Speed'] = 0
df2['Vehicle Speed'] = 0
# Estimated vehicle speeds when braking (AccX < = 0)
df1.loc[df1['AccX'] <= 0, 'Vehicle Speed'] = df1.loc[df1['AccX'] <= 0,
('spd_rpmFL','spd_rpmFR','spd_rpmRL','spd_rpmRR')].max(axis=1)*np.pi/30*r/gear_ratio*3.6
df2.loc[df2['AccX'] <= 0, 'Vehicle Speed'] = df2.loc[df2['AccX'] <= 0,
('spd_rpmFL','spd_rpmFR','spd_rpmRL','spd_rpmRR')].max(axis=1)*np.pi/30*r/gear_ratio*3.6
# Estimated vehicle speeds when accelerating (AccX > 0)
df1.loc[df1['AccX'] > 0, 'Vehicle Speed'] = df1.loc[df1['AccX'] > 0,
('spd_rpmFL','spd_rpmFR','spd_rpmRL','spd_rpmRR')].min(axis=1)*np.pi/30*r/gear_ratio*3.6
df2.loc[df2['AccX'] > 0, 'Vehicle Speed'] = df2.loc[df2['AccX'] > 0,
('spd_rpmFL','spd_rpmFR','spd_rpmRL','spd_rpmRR')].min(axis=1)*np.pi/30*r/gear_ratio*3.6
# Output 62.2 ms ± 3.78 ms per loop (mean ± std. dev. of 7 runs, 10 loops each) Eso fue muy rápido. Como puede ver, si se acostumbra a indexar las tablas MATLAB, el cambio a Pandas Dataframes es mucho más fácil. La sintaxis (o más bien, la lógica) es muy similar, aunque no es exactamente la misma.
Ahora, podemos comparar las velocidades. Primero, calculé los horarios de inicio y finalización para ambos controladores. Esto no es preciso, acabo de definir un umbral de velocidad de 20 km/h y encontré los instantes en que el automóvil era más lento y más rápido que eso. Nuevamente, DataFrame Sleting es muy útil para esto:
# Find index of first instant at which speed is greater than 20 km/h
index_start1 = df1[df1['Vehicle Speed']>20].index[0]
index_start2 = df2[df2['Vehicle Speed']>20].index[0]
time_start1 = df1['Time_s'][index_start1]
time_start2 = df2['Time_s'][index_start2]
# Find index of last instant at which speed is greater than 20 km/h
index_end1 = df1[df1['Vehicle Speed']>20].index[-1]
index_end2 = df2[df2['Vehicle Speed']>20].index[-1]
time_end1 = df1['Time_s'][index_end1]
time_end2 = df2['Time_s'][index_end2]
# Compute elapsed times
t_elap_1 = time_end1 - time_start1
t_elap_2 = time_end2 - time_start2A continuación, hice una trama comparativa (subtramas nuevamente). Sin embargo, esta vez con un enfoque diferente:
# %% Compare the speeds of the two drivers
from plotly.subplots import make_subplots
import plotly.graph_objects as go
# Create figure with subplots and add titles
fig = make_subplots(
rows=2,
cols=1,
shared_xaxes=True,
shared_yaxes=False,
subplot_titles=((f"Driver 1 approximate time : {t_elap_1:.1f} seconds<br>"
f"Max. speed: {df1['Vehicle Speed'].max():.1f} km/h, "
f"mean speed: {df1['Vehicle Speed'].mean():.1f} km/h"),
(f"Driver 2 approximate time : {t_elap_2:.1f} seconds<br>"
f"Max. Speed: {df2['Vehicle Speed'].max():.1f} km/h, "
f"mean speed: {df2['Vehicle Speed'].mean():.1f} km/h")
))
# Plot data for the first driver
fig.add_trace(
go.Line(y=df1['Vehicle Speed'] ,x=df1.Time_s,name = 'Driver 1'),
row = 1, col = 1)
# Add vertical lines to mark start and end points
for time in [time_start1, time_end1]:
fig.add_shape(go.layout.Shape(type = "line",
yref = "y", xref = "x",
x0 = time, y0=0,
x1 = time, y1 = 100),
row=1, col=1)
fig.update_yaxes(title_text="Speed [km/h]", range=[0, 100], row=1, col=1)
# Plot data for the second driver
fig.add_trace(
go.Line(y=df2['Vehicle Speed'] ,x=df1.Time_s,name = 'Driver 2'),
row = 2, col = 1)
# Add vertical lines to mark start and end points
for time in [time_start2, time_end2]:
fig.add_shape(go.layout.Shape(type = "line",
yref = "y", xref = "x",
x0 = time, y0=0,
x1 = time, y1 = 100),
row=2, col=1)
fig.update_yaxes(title_text="Speed [km/h]", range=[0, 100], row=2, col=1)
fig.update_layout(title = {
'text': '<b>Driver time comparison</b>',
'xanchor' : 'center',
'x' : 0.5, 'y': .95, 'font_size' : 20
},
showlegend=False)
figObserve que usé Plotly en lugar
de Pyplot. Nombres confusos, ¿verdad? Pyplot es el "vainilla" que viene
con matplotlib. Es más fácil aprender cuando comienza con Python desde
un fondo de Matlab y se usa mucho para figuras de grado científico y de
ingeniería. Pero Plotly ofrece la posibilidad de hacer tramas
interactivas con un poco de codificación, lo cual es increíble. Solo
mira los resultados:
© 2023 Datapane . Reservados todos los derechos.
Concluir
Completamos la primera parte de la tarea, obteniendo los mismos resultados que el ejemplo de Matlab, ¡afortunadamente! De lo contrario, habría un gran error en alguna parte.
Al escribir esto, creo que pasé el 80% de mi tiempo descubriendo cómo hacer que las parcelas parecieran que quería. El resto no fue realmente tan difícil. Esto no es sorprendente, ya que Python ha ganado popularidad precisamente por ser fácil de aprender. Es fácil, siempre que lo use como está destinado. La mejor manera de lograr esto es seguir un conjunto de pautas para el estilo de codificación, que se conocen como la "pitónica" . Intentaré aprender esto y aplicarlo a mi código a medida que avanza. Creo que te hace pensar más en términos de eficiencia del código y hacer un mejor uso del idioma.
Espero que estos ejemplos de código sean útiles para usted. En la próxima publicación, completaré la segunda parte de la tarea, utilizando histogramas y estadísticas para analizar las velocidades del vehículo y el rendimiento de frenado de los dos conductores.
Mientras tanto, ¡espero que tengas un día maravilloso!

No hay comentarios.:
Publicar un comentario